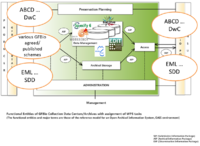
Treffen zu EML- bzw. SDD-strukturierten Daten: Darstellung im GFBio VAT System
Ort
SNSB IT Center München, Menzinger Straße 67, rechter Gebäudeflügel, Parterre, Raum 035
Datum und Zeit
7. Juli 2017, 11.00 Uhr bis 16.30 Uhr
Teilnehmer
Gäste: Thomas Köhler (erkrankt), Gerhard Rambold (Universität Bayreuth, LIAS gtm, NavKey)
Michael Mattig, Bernhard Seeger (Universität Marburg, VAT-Tool)
Anton Link (erkrankt), Dieter Neubacher, Wolfgang Reichert (nachmittags), Veronica Sanz, Stefan Seifert, Dagmar Triebel, Tanja Weibulat (SNSB IT Center, DiversityDescriptions, SDD Data-Pipelines für GFBio, DEEMY)
Protokoll: Tanja Weibulat (für GFBio-Mitarbeiter im internen Wiki zu finden)
Thema
Anhand von publizierten funktionellen und phylogenetischen Trait-Daten (nach EML bzw. SDD strukturiert und als csv oder xml verfügbar) soll die informationstechnische Seite der Visualisierung und Analyse derartiger Datenquellen besprochen werden.
Wegen Terminüberschneidungen organisiert Frau Gerstner ein zweites Treffen in München am 11.7., dann mit Robert Huber und Entwicklern in München.
Sie stellt aber trotzdem Material und Vortrag zu EML für den 7.7. bereit.
Agenda
- ab 10.30 Uhr Eintreffen der Teilnehmer
- 11.00 Begrüßung (D. Triebel)
- 11.05 Uhr Demo des GFBio VAT Tools, mögliche Interoperabilität mit SDD und EML (M. Mattig, B. Seeger)
- 11.45 Uhr Demo von DEEMY Portal mit NaviKey Applet zur Merkmalsauswahl und interaktiven Identifikation (G. Rambold, D. Triebel)
- 12.00 Uhr DEEMY und BEF China, Beispiele für Datenmanagement in DiversityDescriptions (V. Sanz)
- 12.15 Uhr Demo von LIAS gtm, Daten-Export als csv, Erörterung einer möglichen Datenbereitstellung der Exporte für das GFBio VAT Tool (G. Rambold)
- 12.45 Uhr Mittagspause, Café Botanischer Garten
- 13.30 Uhr Datenmanagement mit DiversityDescriptions, Pipelines zum Datenimport z. B. als csv, und Datenexport, Datenpublikation, SDD xml-Archive im GFBio Wiki (DEEMY html, SDD-structured xml, DELTA) (V. Sanz)
- 14.00 Uhr Community Standard SDD wie in DD Namensschema und Community Standard EML (D. Triebel)
- 14.15 Uhr Datenmanagement ökologischer Datenpakete mit Morpho; Datenbereitstellung als EML für GFBio VAT Tool mit MetaCat über SGN Webserver (T. Weibulat in Vertretung von E.-M. Gerstner)
- 14.45 Uhr Diskussion über Möglichkeiten der Datenbereitstellung für GFBio VAT Tool über Daten-Pipelines aus DiversityProjects + DiversityDescriptions-Installationen; GFBio Datenpakete mit GFBio Submission Meta-Information, diese strukturiert nach welchem Community Standard (ABCD, EML, SDD, OAI-PMH, PanSimple)? Daten und Metadaten sollen als xml-Archive angeboten werden über SNSB Webserver? Welche Meta-Informationen braucht das VAT Tool?
- 15.15 Uhr Kaffeepause
- 15.45 Uhr Abschlussdiskussion und angestrebte Kooperationen, inkl. Diskussion über SDD/ EML/ OAI-PMH Mapping und Conversion Tools zwischen Community Standards integriert in DWB Data Pipelines? ToDos - GFBio Phase III?
- 16.30 Uhr Ende
Links
- GFBio intern: GFBio SDD DIPs als xml-Archive
- GFBio intern: EML to PanSimple Mapping
- GFBio intern: Metadata Ingest via DWB Data Pipelines
- GFBio intern: Treffen zu EML- bzw. SDD-strukturierten Daten II
- dwbD database scheme: dwbDescriptions; name space follows the xml-based TDWG standard SDD (=Structured Descriptive Data) as explained in Hagedorn (2008)
References
- Authmann C., Beilschmidt C., Drönner J., Mattig M. & Seeger B. (2015). VAT: A System for Visualizing, Analyzing and Transforming Spatial Data in Science. Datenbank-Spektrum 15(3): 175-184 (doi:10.1007/s13222-015-0197-y)
- Hagedorn, G. (2008/2007). Structuring descriptive data of organisms – Requirement analysis and information models. – 1–417. Dissertation, Universität Bayreuth.
- Hagedorn, G., Plank, A., Link, A., Rambold, G. & Triebel, D. (2016). DiversityDescriptions information model (version 3.0.15, 11 July 2016). http://www.diversityworkbench.net/Portal/DiversityDescriptionsModel_3.0.15.
- Rambold, G., Zedda, L., Coyle, J., Peršoh, D., Köhler, T., Triebel, D. (2016). Geographic heat maps of lichen traits derived by combining LIAS light description and GBIF occurrence data, provided on a new platform. – Biodiversity and Conservation 25(13): 2743–2751. (doi:10.1007/s10531-016-1199-2)